今回はDataFrameの処理速度を
向上させるためのコツについて解説します
この記事を読むべき方・・・
- Pythonでデータ分析や機械学習を行うエンジニア
- 大量データを扱い、処理速度に課題を感じているユーザー
- Pandasを使いこなしたいと考えている初級〜中級者
コツ1: ベクトル化を使った効率的なデータ処理
for文などを使用せずに、行や列に対して
一括処理を行うことで処理速度を向上させます
処理速度が遅いよー
今回は例として
iterrowsを使用したコードと
ベクトル化を使用したコードを比較します
タイムの計測には%%timeitを使用します
Before: iterrowsを使用したコード
itterowsを使用して
A列とB列の値を足し合わせた,C列を作成します
import pandas as pd
import numpy as np
import time
# データの準備(A, B列にそれぞれランダムな値がある10万行のデータフレームを作成)
df = pd.DataFrame({'A': np.random.rand(100000), 'B': np.random.rand(100000)})
# データの確認
print(df)
A B
0 0.055674 0.321138
1 0.246434 0.251720
2 0.635473 0.411995
3 0.517269 0.957901
4 0.382983 0.708736
... ... ...
99995 0.185137 0.689433
99996 0.552369 0.492906
99997 0.208750 0.157748
99998 0.296905 0.114558
99999 0.991895 0.257504
# iterrowsを使用した処理
%%timeit
df['C'] = np.nan
for index, row in df.iterrows():
df.at[index, 'C'] = row['A'] + row['B']
# 処理時間
26.8 s ± 641 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
After: ベクトル化を使用したコード
iterrowsと同様の処理を
ベクトル化を使用して行います
# ベクトル化を使用した処理
%%timeit
df['C'] = df['A'] + df['B']
# 処理時間
1.33 ms ± 423 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
処理速度は以下のとおりでした
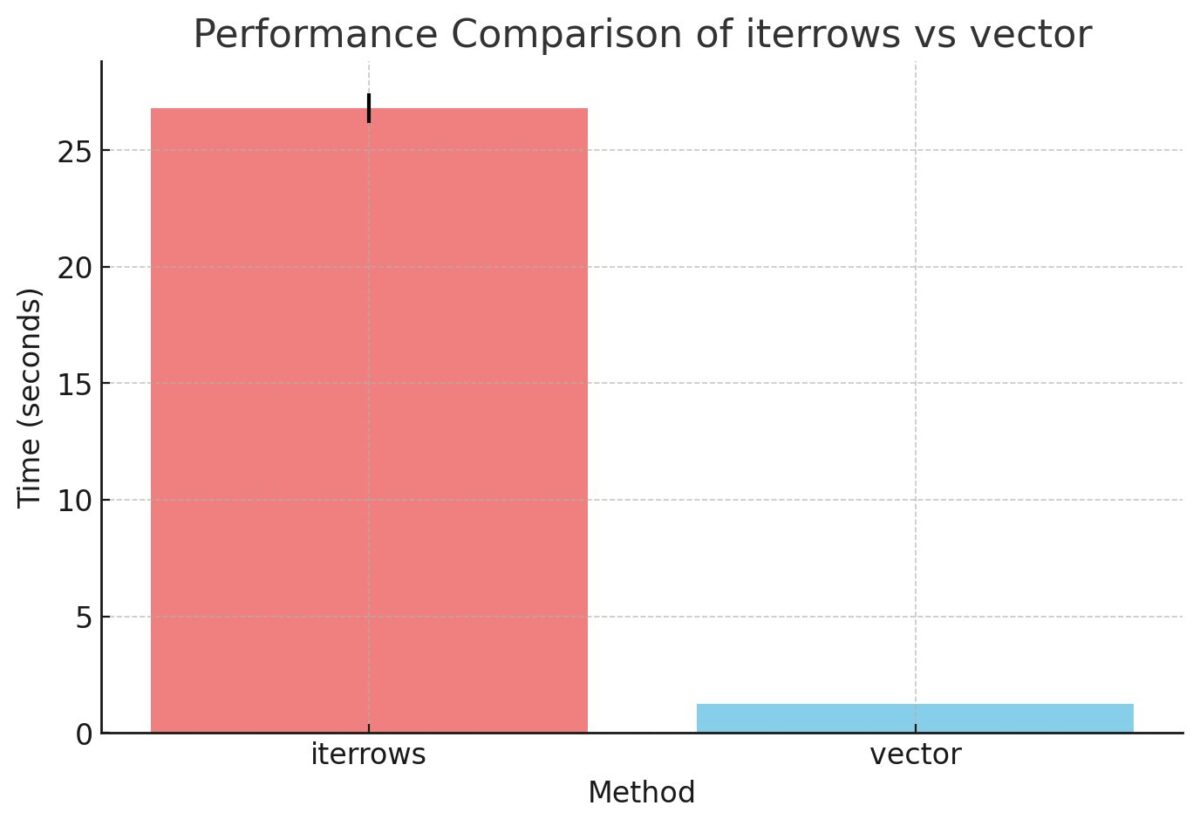
ベクトル化の方が圧倒的に速い結果となりました
高速化の理由・・・
ベクトル化は行ごとの処理ではなく
列を一括で処理するため処理速度が大幅に向上します
コツ2: データ型を最適化してメモリ使用量を削減
データ型の最適化は
メモリ使用量の削減と
処理速度の改善に直結します
今回の例では以下の2つの例を紹介します
- float64型 → float32型
- object型 → category型
float64からfloat32への変換
Before: float64型の使用
例として10万行のデータフレームを作成し
A列とB列の値を足し合わせた,C列を作成します
import pandas as pd
import numpy as np
import time
# データの準備(A, B列にそれぞれランダムな値がある10万行のデータフレームを作成)
df = pd.DataFrame({'A': np.random.rand(100000), 'B': np.random.rand(100000)})
# データの確認
print(df)
A B
0 0.104770 0.460523
1 0.451645 0.743672
2 0.415677 0.348527
3 0.089152 0.404354
4 0.279895 0.849563
... ... ...
99995 0.400840 0.408237
99996 0.299817 0.691443
99997 0.427719 0.217992
99998 0.910969 0.620087
99999 0.200447 0.234848
[100000 rows x 2 columns]
# データフレームの情報を表示(float64型であることを確認)
print(df.info())
Data columns (total 2 columns):
# Column Non-Null Count Dtype
0 A 100000 non-null float64 # データ型がfloat64であることを確認
1 B 100000 non-null float64 # データ型がfloat64であることを確認
dtypes: float64(2)
memory usage: 1.5 MB # メモリ使用量が1.5MB
# 処理速度の計測
%%timeit
df['C'] = df['A'] + df['B']
# データの確認(処理後)
print(df)
A B C
0 0.104770 0.460523 0.565293
1 0.451645 0.743672 1.195317
2 0.415677 0.348527 0.764205
3 0.089152 0.404354 0.493506
4 0.279895 0.849563 1.129458
... ... ... ...
99995 0.400840 0.408237 0.809077
99996 0.299817 0.691443 0.991260
99997 0.427719 0.217992 0.645711
99998 0.910969 0.620087 1.531056
99999 0.200447 0.234848 0.435295
[100000 rows x 3 columns]
# 処理時間
1.2 ms ± 225 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
After: float32型に変換
float64型と同様の処理を
float32型に変換して行います
# float32型への変換
df['A'] = df['A'].astype('float32')
df['B'] = df['B'].astype('float32')
# データフレームの情報を表示(float32型であることを確認)
print(df.info())
Data columns (total 2 columns):
# Column Non-Null Count Dtype
0 A 100000 non-null float32 # データ型がfloat32に変換されていることを確認
1 B 100000 non-null float32 # データ型がfloat32に変換されていることを確認
dtypes: float32(2)
memory usage: 781.4 KB # メモリ使用量がfloat64の半分になっている
# 処理速度の計測
%%timeit
df['C'] = df['A'] + df['B']
# 処理時間
633 µs ± 168 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
処理速度は以下のとおりでした
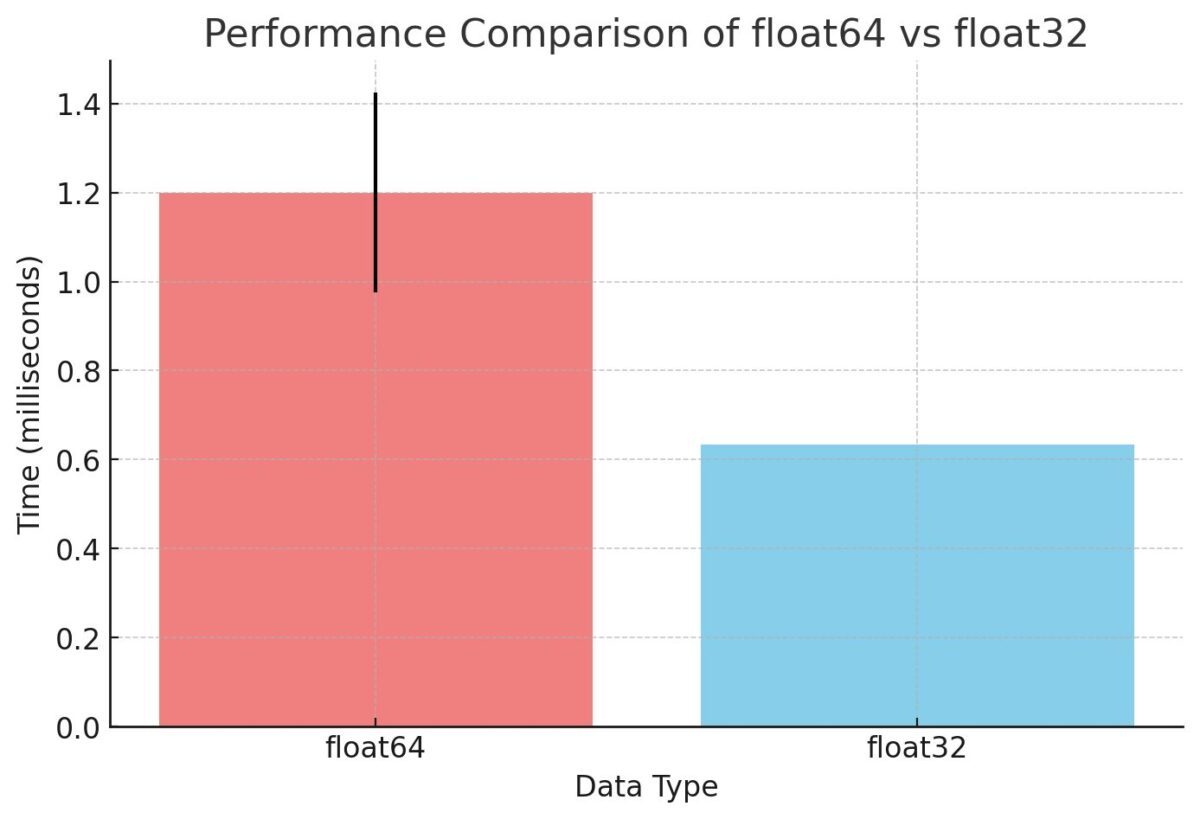
float32の方が2倍程度速い結果となりました
高速化の理由・・・
float32の方が表現できる桁数が少ない分
メモリ使用量も少なく処理も速くなります
df.info()の結果をみてもメモリ使用量が
1.5MB(float64)から781.4KB(float32)に
削減されていることがわかります
float64とfloat32の違いを以下に
簡単に説明しておくので
気になる方はクリックして確認してください
- float64とfloat32の違いはこちらをクリック
float64
- 64ビット(8バイト)で表現される浮動小数点数
- 精度が高い(約15〜16桁の精度)
- メモリ使用量が多い
- 数値範囲が広い
float32
- 32ビット(4バイト)で表現される浮動小数点数
- 精度が低い(約7桁の精度)
- メモリ使用量が少ない
- 数値範囲が狭い
float32/float64の使い分け
- float32:精度があまり必要がなく処理速度を重視する場合
- float64:より高い精度が求められる科学計算をする場合など
objectからcategoryへの変換
Before: object型を使用したコード
例として20万行の都市名が入ったcity列を作成し
city列を大文字に変換したcity_upper列を作成します
# データの準備(都市名を格納した10万行のデータフレームを作成)
df = pd.DataFrame({'city': ['Tokyo', 'Osaka', 'Nagoya', 'Tokyo', 'Osaka'] * 200000})
# データの確認
print(df)
city
0 Tokyo
1 Osaka
2 Nagoya
3 Tokyo
4 Osaka
... ...
999995 Tokyo
999996 Osaka
999997 Nagoya
999998 Tokyo
999999 Osaka
[1000000 rows x 1 columns]
# データフレームの情報を表示(object型であることを確認)
print(df.info())
Data columns (total 1 columns):
# Column Non-Null Count Dtype
0 city 1000000 non-null object # データ型がobjectであることを確認
dtypes: object(1)
memory usage: 7.6+ MB # メモリ使用量が7.6MB
# 処理速度の計測
%%timeit
df['city_upper'] = df['city'].str.upper()
# データの確認(処理後)
print(df)
city city_upper
0 Tokyo TOKYO
1 Osaka OSAKA
2 Nagoya NAGOYA
3 Tokyo TOKYO
4 Osaka OSAKA
... ... ...
999995 Tokyo TOKYO
999996 Osaka OSAKA
999997 Nagoya NAGOYA
999998 Tokyo TOKYO
999999 Osaka OSAKA
[1000000 rows x 2 columns]
# 処理時間
872 ms ± 172 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
After: category型に変換
object型と同様の処理を
category型に変換して行います
# category型への変換
df['city'] = df['city'].astype('category')
# データフレームの情報を表示(category型であることを確認)
print(df.info())
Data columns (total 1 columns):
# Column Non-Null Count Dtype
0 city 1000000 non-null category # category型に変換
dtypes: category(1)
memory usage: 976.8 KB # メモリ使用量が7.6MBから976.8KBに削減
# 処理速度の計測
%%timeit
df['city_upper'] = df['city'].str.upper()
# 処理時間
30.5 ms ± 4.59 ms per loop (mean ± std. dev. of 7 runs, 10 loops each)
処理速度は以下のとおりでした
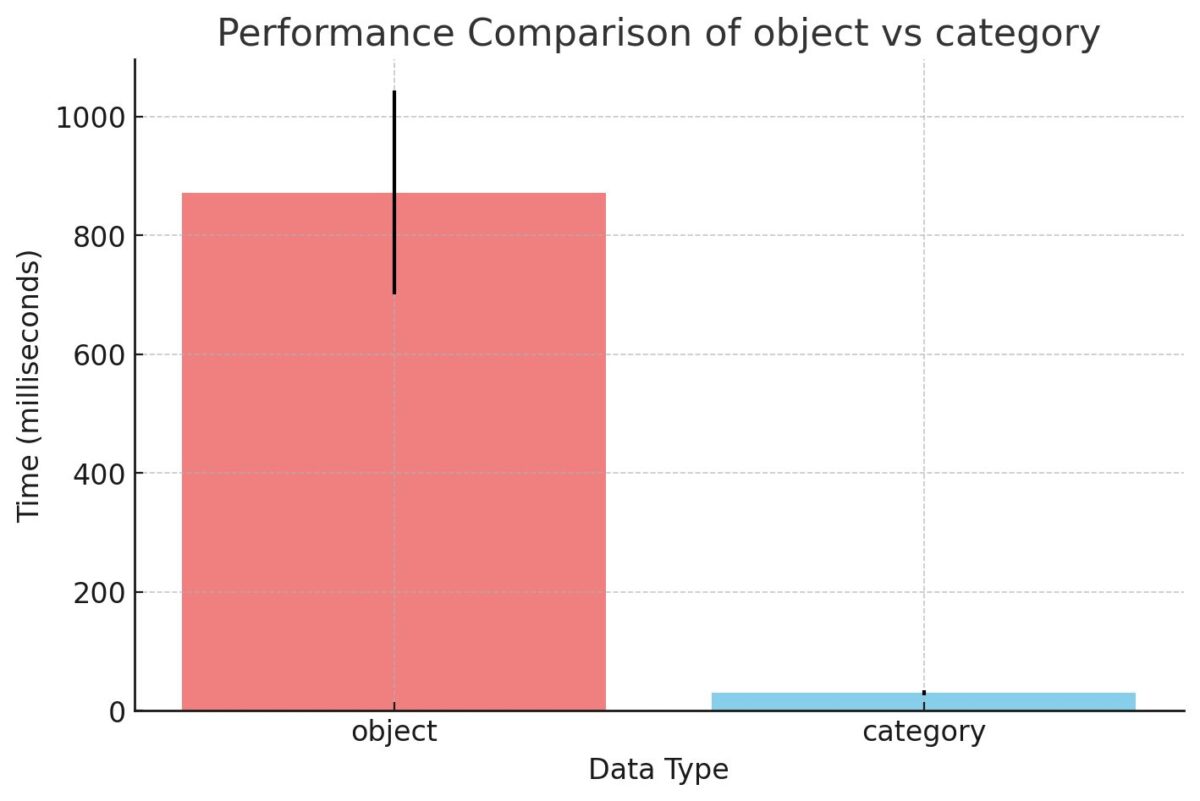
category型の方が圧倒的に速い結果となりました
高速化の理由・・・
カテゴリ型は内部的に整数でデータを扱うため
文字列よりも比較や検索
ソートなどの操作が高速に行えます
df.info()の結果をみてもメモリ使用量が
7.6MB(object)から976.8KB(category)に
削減されていることがわかります
object型とcategory型の違いを以下に
簡単に説明しておくので
気になる方はクリックして確認してください
- object型とcategory型の違いはこちらをクリック
オブジェクト型 (object)
- 説明: 文字列(テキスト)や混合データ型を扱うためのデータ型
- 用途例: 名前や住所、説明などのテキストデータを保存する
- 特徴:メモリ消費→多,処理速度→遅
カテゴリ型 (category)
- 説明: 限られた数のユニークな値(カテゴリ)を持つデータ型
- 用途例: 性別(男性/女性)や、状態(OK/NG)などのカテゴリデータを効率的に保存する
- 特徴:メモリ消費→少, 処理速度→速
比較
- メモリ効率: カテゴリ型 > オブジェクト型
- パフォーマンス: カテゴリ型 > オブジェクト型
コツ3: NumPyのみを使用した計算を使用する
Pandasの内部で使用されるNumPyを
直接使用して処理速度を向上できます
Before: Pandasを使った計算
100行のデータフレームを作成し
A列の値を2乗したB列を作成します
import pandas as pd
import numpy as np
import time
# データの準備(A列にランダムな値がある100万行のデータフレームを作成)
df = pd.DataFrame({'A': np.random.rand(1000000)})
# データの確認
print(df)
A
0 0.394342
1 0.195238
2 0.966231
3 0.831709
4 0.634390
... ...
999995 0.261430
999996 0.123959
999997 0.505840
999998 0.112474
999999 0.727452
1000000 rows × 1 columns
# 処理速度の計測(2乗計算)
%%timeit
df['B'] = df['A'] ** 2
# データの確認(処理後)
print(df)
A B
0 0.394342 0.155505
1 0.195238 0.038118
2 0.966231 0.933601
3 0.831709 0.691740
4 0.634390 0.402451
... ... ...
999995 0.261430 0.068345
999996 0.123959 0.015366
999997 0.505840 0.255874
999998 0.112474 0.012650
999999 0.727452 0.529186
1000000 rows × 2 columns
# 処理時間
6.1 ms ± 837 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
After: NumPyを使用した計算`
NumPyを使用して
Pandasと同様の処理を行います
# 処理速度の計測(2乗計算)
%%timeit
df['B'] = np.square(df['A'].values)
# 処理時間
4.33 ms ± 363 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
処理速度は以下のとおりでした
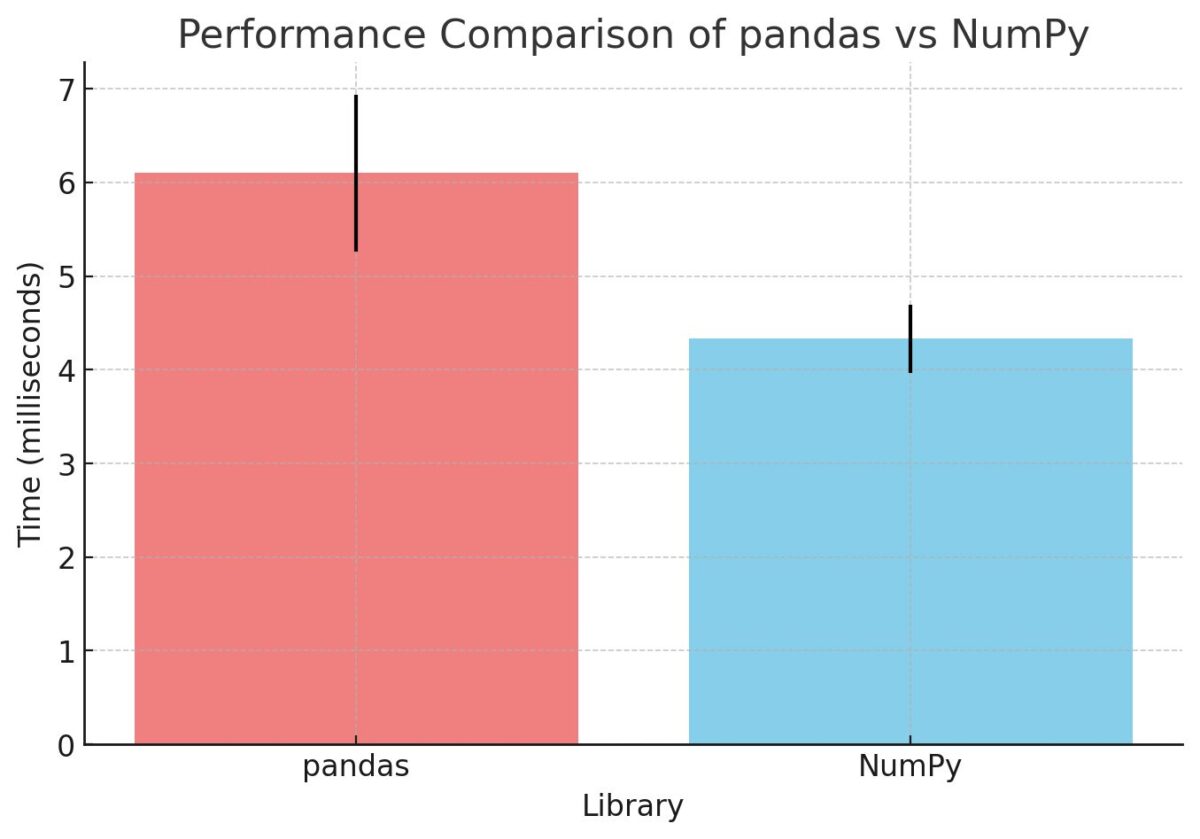
NumPyの方が少しだけ速い結果となりました
高速化の理由・・・
numpyは内部的にC言語で実装されているため
高速な計算が可能です
NumbaによるJITコンパイルの活用
この方法については別の記事で
詳しく解説しているので
参考にしてみてください
処理速度をupさせると
精度の低下などのデメリットもあるため
メリット・デメリットを把握した上で
使用することが大切です!


